Resumo
Revista Brasileira de Ginecologia e Obstetrícia. 2015;37(5):203-207
DOI 10.1590/SO100-720320150005293
Determinar a frequência do Papilomavírus Humano (HPV) na placenta, no colostro e no sangue do cordão umbilical de parturientes e seus neonatos atendidos no Ambulatório de Ginecologia e Obstetrícia do Hospital Universitário de Rio Grande (RS), Brasil.
Foram coletadas biópsias de 150 placentas do lado materno, 150 do lado fetal, 138 amostras do sangue do cordão umbilical e 118 amostras de colostro. As biópsias de placenta foram coletadas da porção central e periférica. O DNA foi extraído segundo protocolo do fabricante e conforme referência encontrada na literatura. O HPV foi detectado pela técnica da reação em cadeia da polimerase aninhada (PCR-Nested) com os primers MY09/11 e GP5/GP6. A genotipagem foi por sequenciamento direto. As participantes responderam a um questionário autoaplicado com dados demográficos e clínicos, a fim de caracterizar a amostra.
O HPV foi detectado em 4% (6/150) do lado materno das placentas, 3,3% (5/150) do lado fetal; 2,2% (3/138) no sangue do cordão e 0,8% (1/118) no colostro. A taxa de transmissão vertical foi de 50%. O genótipo de baixo risco oncogênico encontrado foi o HPV-6 (60%) e de alto risco, os HPV-16 e HPV-18 (20% cada).
Esses resultados sugerem que o HPV pode infectar a placenta, o colostro e o sangue do cordão umbilical.
Resumo
. ;:94-99
DOI 10.1590/SO100-720320150005206
Comparar o desempenho de duas técnicas de genotipagem de papilomavírus humano (HPV), Linear Array e PapilloCheck, em mulheres com lesão intra-epitelial de alto grau (LIAG).
Foram selecionadas 88 mulheres com diagnóstico citopatológico de LIAG em 2 centros de referência em patologia cervical em Salvador, Bahia, no período de julho de 2006 a janeiro de 2009. Após o diagnóstico citopatológico de LIAG, foram realizadas a coleta de células do colo uterino para a genotipagem do HPV e a biópsia sob visão colposcópica para análise histopatológica do fragmento retirado. Posteriormente à confirmação de NIC2+ pelo exame histopatológico, foi realizada a genotipagem do HPV em 41 mulheres pelas técnicas Linear Array e PapilloCheck.
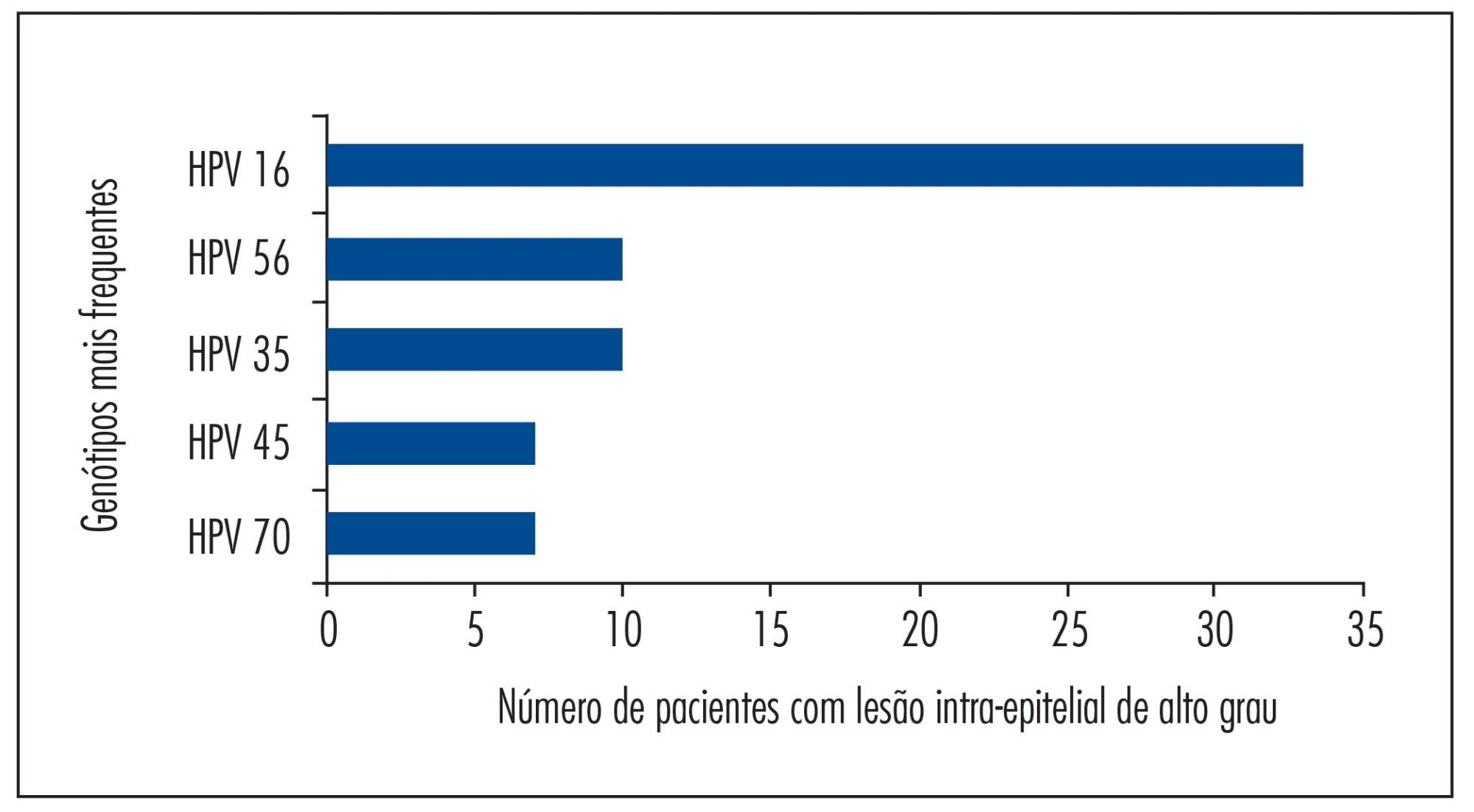
Os dois testes apresentaram taxa de concordância geral para detecção do vírus HPV de 97,2% (35/36). Das 36 amostras válidas, 35 (97,2%) foram consideradas positivas em ambos os testes e apenas uma amostra (2,8%) foi considerada discordante. Os genótipos do HPV mais prevalentes detectados através da técnica do Linear Array foram: HPV 16, HPV 56, HPV 35, HPV 45 e HPV 70; e pela técnica PapilloCheck foram: HPV 16, HPV 56, HPV 11, HPV 35 e HPV 42. Foi observado índice semelhante de infecção por múltiplos tipos do HPV nos dois testes analisados (72,5% no Linear Array e 75,0% no PapilloCheck).
Os testes de genotipagem Linear Array e PapilloCheck apresentaram um desempenho equivalente na detecção dos tipos de HPV oncogênicos em mulheres com LIAG, tendo o PapilloCheck a vantagem de ser um método que evita a subjetividade da leitura dos genótipos de HPV.
Resumo
. ;:416-422
DOI 10.1590/SO100-720320140004995
O objetivo foi descrever a distribuição dos genótipos do papilomavírus humano e a frequência de infecções por múltiplos genótipos, bem como avaliar a associação entre genótipos de papilomavírus humano, faixa etária e resultados cito-histopatológicos.
Estudo retrospectivo de corte transversal realizado entre junho de 2010 e outubro de 2013 em Salvador, Bahia, Brasil. Foram revisados 351 prontuários de mulheres com genotipagem positiva pelo teste PapilloCheck(r), usado para detectar 24 tipos de papilomavírus humano. Os achados cito-histopatológicos foram classificados em grupos de: achados negativos para neoplasia (exames citopatológico e histopatológico negativos), lesão de baixo grau (achado citopatológico - lesão intraepitelial de baixo grau - ou achado histopatológico - neoplasia intraepitelial cervical grau 1, neoplasia intraepitelial vaginal grau 1 ou condiloma e lesão de alto grau (achado citopatológico - lesão intraepitelial de alto grau - ou histopatologia com laudo maior ou igual a neoplasia intraepitelial cervical grau 2 ou neoplasia intraepitelial vaginal grau 2).
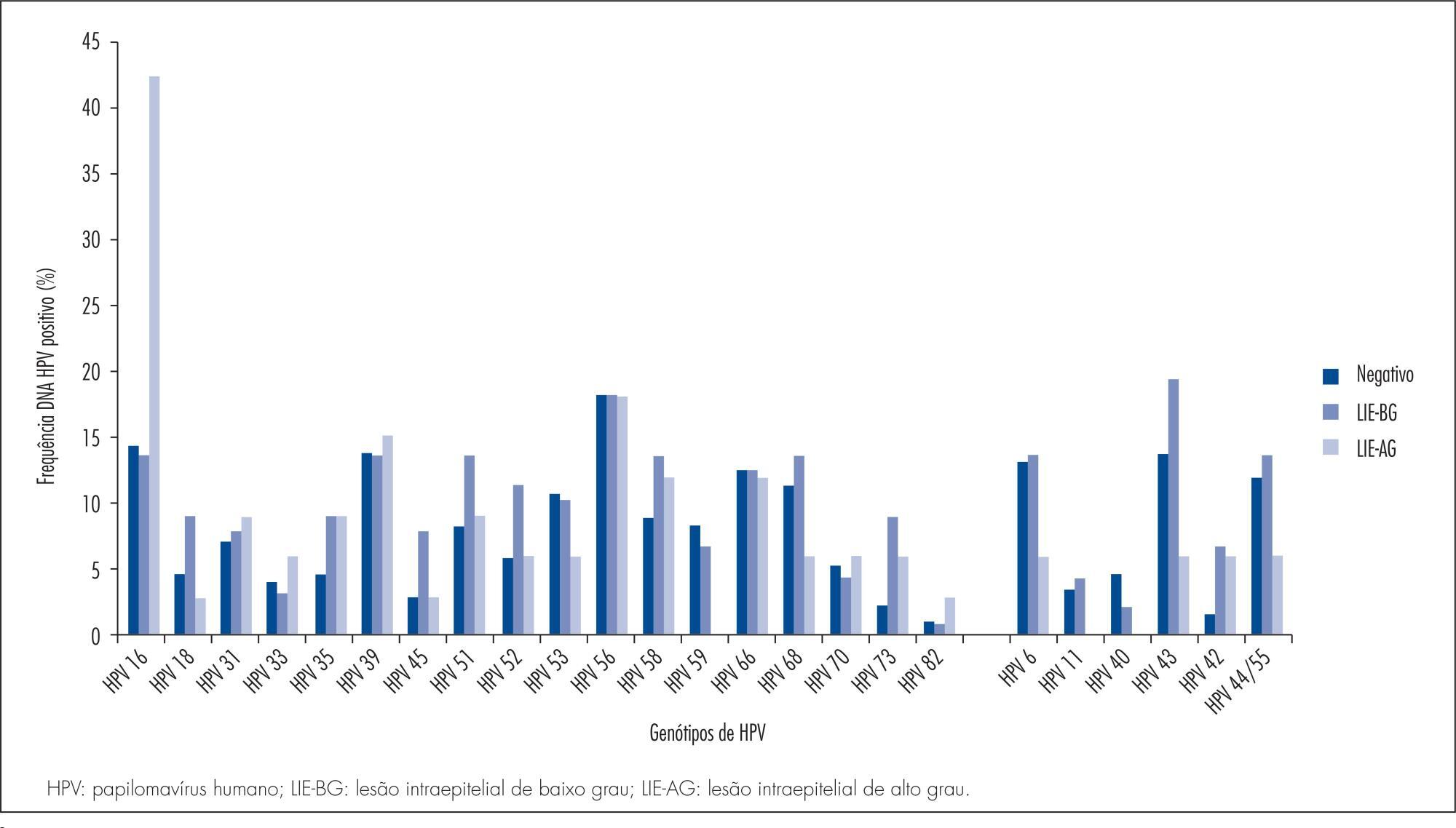
O genótipo de alto risco mais frequente foi o HPV 16, com 18,5%; intervalo de confiança de 95% (IC95%) 14,6-23,0, seguido pelo HPV 56 (14%; IC95% 10,5-18,0) e o HPV 39 (13,4%; IC95% 9,5-16,8). O HPV 18 (5,4%; IC95% 3,3-8,3) esteve entre os menos comuns. Entre os tipos de baixo grau, o HPV 42 (15,7%; IC 95% 12,0-20,0), o HPV 6 (11,4%; IC95% 8,3-15,2) e o HPV 44/55 (11,1%; IC95% 8,0-14,9) foram os mais encontrados, enquanto o HPV 11 (2,8%; IC95% 1,4-5,2) foi o menos frequente. A proporção do HPV 16 aumentou com a severidade das anormalidades cito-histopatológicas de 13,8% (12/87) nas lesões de baixo grau para 42,4% (14/33) nas lesões de alto grau. Houve associação significativa entre a presença de lesão cito-histopatológica de baixo ou alto grau e os genótipos de alto risco, HPV 16, HPV 52, HPV 73 e HPV 82, e o de baixo risco, HPV 43. Mulheres com menos de 30 anos apresentaram frequência significativamente maior do HPV 16 (22,2 versus 12,9%, p=0,01), do HPV 42 (19,7 versus 10,9%, p=0,01) e do HPV 45 (6,6 versus 1,4%, p=0,01), além de infecção múltipla (58,1 versus 47,4%, p=0,04).
Observou-se uma variabilidade da distribuição dos diversos genótipos de papilomavírus humano em mulheres no estado da Bahia. Na amostra estudada, o HPV 16 foi o mais frequente, assim como em outras regiões do Brasil e do mundo. Encontramos o HPV 56 e o HPV 39 como o segundo e o terceiro mais frequentes. Entretanto, o HPV 18 esteve entre os menos comuns. Os tipos não oncogênicos, HPV 42, 6 e 44/55, foram os mais observados, enquanto o HPV11 foi o menos frequente.
Resumo
Revista Brasileira de Ginecologia e Obstetrícia. 2011;33(10):315-320
DOI 10.1590/S0100-72032011001000008
OBJETIVO: comparar três métodos para detecção do HPV e determinar a prevalência dos genótipos encontrados. MÉTODOS: um total de 120 amostras de raspagem da região cervical de mulheres portadoras de neoplasia intraepitelial cervical foram analisadas pela reação em cadeia da polimerase convencional, usando os sistemas de primers MY09/11, GP05+/06+ e pela Nested-PCR. As amostras foram submetidas à extração de DNA e, logo após, amplificadas com os primers GH20 e PC04 (β-globina) para verificação da qualidade do DNA obtido e pela reação em cadeia da polimerase convencional e Nested-PCR. Os fragmentos amplificados foram visualizados em gel de agarose a 1,2%, corados com Blue Green Loading Dye I. As amostras positivas foram sequenciadas usando o sequenciador automático de DNA "MegaBACE 1000". Para análise estatística foram utilizados os teste do Χ2 e o de Fisher com nível de significância de 5%. RESULTADOS: quinze amostras não se amplificaram para os primers de β-globina, sendo eliminadas do estudo. Das amostras restantes, 40% (42/105) foram positivas para os primers MY09/11, 98% (103/105) para os primers GP05+/06+ e 92% (97/105) para Nested-PCR. Considerado as técnicas MY09/11 e GP05+/06+, foi possível observar 100% de amostras positivas para o HPV. Neste estudo, a prevalência dos genótipos foi de 58, 23, 5, 4 e 3% para HPVs 16, 18, 31, 33 e 56, respectivamente. Os HPV 67 e 83 apresentaram 2% e os HPV 6, 11, 58 e candHPV85, 1% cada. A prevalência dos genótipos neste estudo está de acordo com o reportado em todo o mundo (IC95%=0,4657-0,8976). CONCLUSÕES: para obter resultados mais confiáveis, é necessário o uso de mais que um sistema de primers para detecção do HPV. Acredita-se que as três técnicas estudadas são importantes e adequadas para o diagnóstico clínico do HPV quando apropriadamente combinadas.
Resumo
Revista Brasileira de Ginecologia e Obstetrícia. 2011;33(2):65-69
DOI 10.1590/S0100-72032011000200002
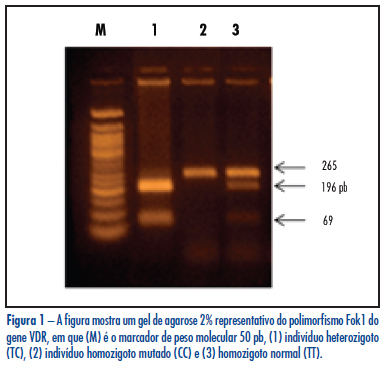
OBJETIVO: avaliar a frequência do polimorfismo Fok1 do gene do receptor da vitamina D (VDR) em mulheres inférteis com endometriose e Grupo Controle, e sua associação com a doença. MÉTODOS: estudo caso-controle que incluiu 147 mulheres inférteis com endometriose e 154 mulheres férteis sem endometriose como Controle. O polimorfismo Fok1 (rs10735810, T2C), que promove uma troca de T/C no éxon 2 do gene VDR, foi identificado por PCR-RFLP (análise de polimorfismos de fragmentos de restrição), que envolve a combinação de amplificação por PCR (reação em cadeia da polimerase) e digestão com endonuclease de restrição. O teste do χ2 foi utilizado para comparar as frequências dos genótipos e alelos entre os grupos. Todos os valores de p foram bicaudais, e o nível de significância considerado foi 0,05 (α<0,05). RESULTADOS: os genótipos TT, TC e CC do polimorfismo FokI do gene VDR apresentaram frequência de 44,2%, 46,9% e 8,9% nas mulheres inférteis com endometriose e 41,6%, 50% e 8,4% no Grupo Controle, e não apresentaram diferença significante quando comparados (p=0,8), mesmo quando as pacientes com endometriose foram subdivididas de acordo com o estadiamento da endometriose (p=0,3 para endometriose mínima e leve e p=0,2 para endometriose moderada e grave). Em relação aos alelos, T e C estavam presentes, respectivamente, em 67,6% e 32,3% das mulheres inférteis com endometriose (p=0,8), em 63,5% e 36,5% das mulheres com endometriose mínima/leve (p=0,5), em 72,5% e 27,5% das mulheres com endometriose moderada/grave (p=0,2), e em 66,6% e 33,4% das mulheres do Grupo Controle. Não foi observada diferença estatística significante na comparação entre nenhum dos grupos e o Grupo Controle. CONCLUSÃO: o estudo mostrou que o polimorfismo Fok1 do gene VDR não confere risco genético associado ao desenvolvimento de endometriose associada à infertilidade na população brasileira.
Resumo
Revista Brasileira de Ginecologia e Obstetrícia. 2010;32(10):476-485
DOI 10.1590/S0100-72032010001000002
OBJETIVO: analisar, em mulheres com HPV em colo do útero, as características da infecção viral e os fatores de risco para lesão intraepitelial de alto grau e carcinoma cervical. MÉTODOS: realizou-se um estudo caso-controle com mulheres com HPV em colo do útero atendidas em serviço de Ginecologia de referência vinculado ao SUS, em Recife, Nordeste do Brasil. No grupo de casos (72 mulheres com lesão intraepitelial de alto grau ou carcinoma cervical) e de controles (176 mulheres com colpocitologia normal ou com alterações benignas), foram pesquisados seis genótipos virais (HPV 16, 18, 31, 33, 6 e 11) em material da ecto- e endocérvice com primers MY09/MY11. As variáveis independentes foram hierarquizadas em três níveis de determinação: distal (sociodemográficas), intermediário (comportamentais) e proximal (realização anterior de colpocitologia). A homogeneidade das proporções foi testada (χ2). Obtiveram-se ORs não ajustadas e, na modelagem final, realizou-se regressão logística hierarquizada com o ajuste do efeito de cada variável sobre o desfecho pelas variáveis do mesmo nível e de níveis anteriores de causalidade. RESULTADOS: em 76,6% das 248 mulheres participantes do estudo, o genótipo viral da infecção cervical foi identificado. Predominaram genótipos de alto risco oncogênico (83,4% nos casos e 67,1% nos controles), principalmente HPV 16 e 31. Foram identificados como fatores de risco (a) distais: residir em zona rural (OR=2,7; IC95%: 1,1-6,2), menos de três anos de estudo (OR=3,9; IC95%: 2,0-7,5) e renda familiar inferior a dois salários mínimos (OR=3,3; IC95%: 1,0-10,5); (b) intermediário: número de gestações igual ou superior a quatro (OR=2,0; IC95%: 1,0-3,7); (c) proximal: ausência de colpocitologia anterior (OR=9,7; IC95%: 2,4-38,2). CONCLUSÕES: em mulheres usuárias do SUS do Nordeste do Brasil predominam os genótipos virais 16 e 31 em infecções cervicais por HPV, sendo que fatores socioeconômicos, reprodutivos e relacionados à ausência de rastreamento citológico representam risco para lesão intraepitelial de alto grau e câncer cervical.
Resumo
Revista Brasileira de Ginecologia e Obstetrícia. 2009;31(12):609-614
DOI 10.1590/S0100-72032009001200006
OBJETIVO: descrever a diversidade genética dos isolados de HIV-1 de mulheres soropositivas acompanhadas em um centro de referência. MÉTODOS: estudo transversal, no qual foram incluídas 96 mulheres com dois testes sorológicos ELISA e um teste confirmatório Western Blot. Das amostras de sangue periférico, foram determinadas a carga viral pelo kit b-DNA e a contagem de linfócitos T CD4 e T CD8 pela citometria de fluxo excalibur. A extração e purificação do DNA pró-viral foi realizada pela reação em cadeia da polimerase (PCR), utilizando o kit QIAamp Blood (Qiagen Inc., Chatsworth, CA, USA). O sequenciamento da região pol foi realizado em 52 isolados com o (3100 Genetic Analyzer, Applied Biosystems Inc., Foster City, CA) e a genotipagem foi investigada pela ferramenta Rega (Rega Subtyping Tool). O padrão de resistência aos antirretrovirais (ARV) foi inferido pelo algoritmo do banco de dados Stanford HIV Resistance. Os estágios clínicos das participantes foram definidos como A, B ou C segundo os critérios do Center for Diseases Control (CDC). Para a análise estatística dos dados, foram utilizados os testes do χ2 para as variáveis categóricas e o teste t de Student para as variáveis numéricas. RESULTADOS: a média de idade da amostra, o tempo médio de doença e de tratamento foram: 33,7; 3,8 e 2,5 anos, respectivamente. A média da carga viral foi log10 2,3 cópias/mL; a dos linfócitos T CD4 e T CD8 foi 494,9 células/µL e 1126,4 células/µL. Sobre o estágio clínico, 30 mulheres estavam no estádio A, 47 no B e 19 no C. O sequenciamento dos 52 isolados encontrou 33 do subtipo B, quatro do F, um do C e 14 do recombinante BF. A análise da resistência aos ARV mostrou 39 (75,0%) isolados susceptíveis, 13 (25,0%) resistentes aos inibidores da transcriptase reversa (INTR) e três (5,7%) aos inibidores da protease (IP). CONCLUSÕES: Houve grande diversidade do HIV-1 e elevado percentual de isolados resistentes aos ARV na amostra estudada.